Artigo sobre o efeito do contra-íon colina sobre o potencial eletrostático de micelas publicado no JMM
março 12, 2024 Deixe um comentário
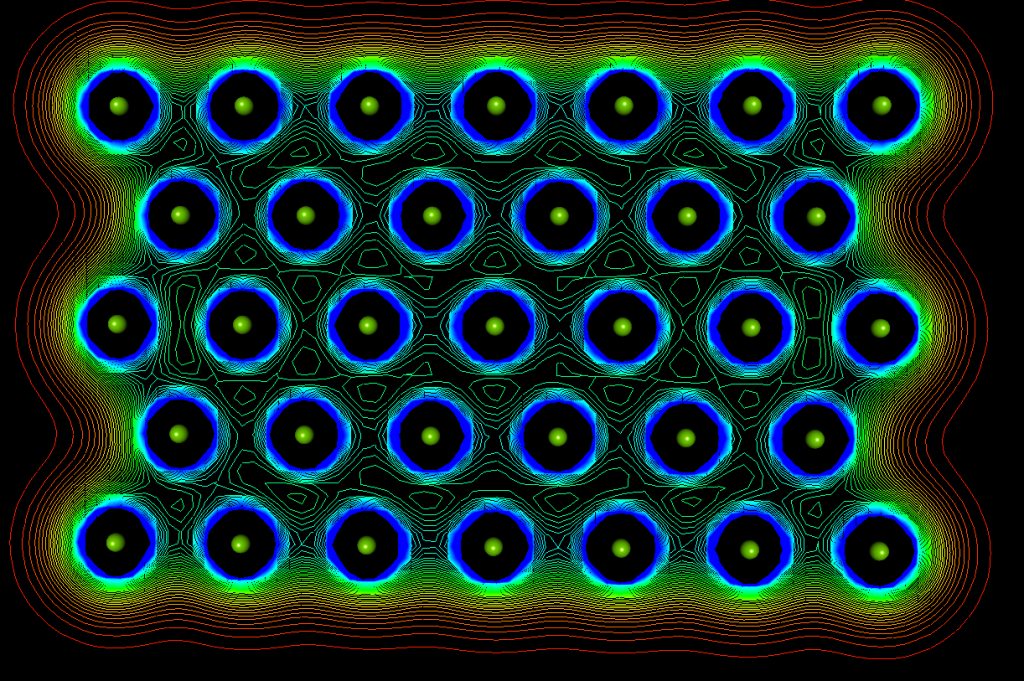
O artigo “Counter-ion adsorption and electrostatic potential in sodium and choline dodecyl sulfate micelles – a molecular dynamics simulation study”, de autoria minha e de Rafaela Eliasquevici, foi publicado no Journal of Molecular Modeling.
Nesse trabalho mostramos como a substituição do contra-íon sódio por colina afeta a forma, a área de superfície acessível ao solvente e o potencial eletrostático de micelas de dodecil-sulfato por meio de simulações de dinâmica molecular clássicas feitas com o programa GROMACS. Foi observado que o contra-íon colina apresenta maior adsorção sobre a superfície da micela, podendo interagir simultaneamente com várias cabeças de surfactante, o que reduz a área exposta de micela à água e torna o potencial de superfície menos negativo. Observou-se também que, apesar da colina poder doar ligação de hidrogênio para o surfactante, a interação mais importante é a interação iônica com o grupo amônio.
O artigo pode ser acessado pelo endereço https://doi.org/10.1007/s00894-024-05897-1 . A versão de autor revisada pode ser vista abaixo: