Tutorial Gromacs – água pura – parte 1
Tutorial Gromacs – água pura parte 1
Nesse tutorial realizaremos uma simulação de dinâmica molecular de uma caixa de água com o programa Gromacs. Esse tutorial tem caráter introdutório, apresentando desde a instalação dos programas a serem utilizados passando pela preparação da caixa de simulação, visualização gráfica da mesma e realização de uma minimização de energia até a realização da simulação propriamente dita. Diversos comentários serão feitos sobre os programas utilizados e arquivos de input, mas para quem quiser partir diretamente para a preparação e execução das simulação sem explicações pode ir para a “versão receita de bolo” ao final dessa página. Na segunda página do tutorial serão abordadas diferentes ferramentes de análise, incluindo ferramentas do próprio Gromacs e do programa Travis. Todos os softwares utilizados aqui são gratuitos para uso não-comercial.
Esse tutorial foi preparado para execução em ambiente Linux e testado nas versões do Gromacs 2018 e 2020. Uma versão mais antiga desse tutorial, que funciona com versões mais antigas do Gromacs, pode ser acessada aqui: https://kalilbn.wordpress.com/dinamica-molecular-conceitos-fundamentais/tutorial-gromacs-agua-pagina-1/
Arquivos necessários para o tutorial: https://drive.google.com/drive/folders/136WPXqb9bhsvjwUHkhEgag8NA29QNhGi?usp=share_link
Conteúdo:
Página 1
- Instalação dos programas utilizados
- Criação e visualização da caixa de simulação
- Minimização de energia
- Execução da simulação
- Receita de bolo (tutorial sem explicações)
.
Página 2 (ir para a página)
- Componentes de energia, densidade, volume, temperatura e pressão
- Ligações de hidrogênio
- Distribuição radial de pares com o Gromacs
- Distribuição radial de pares e distribuição espacial com o Travis
- Problemas propostos
- Referências
.
1. Instalação dos programas utilizados
Para a primeira parte desse tutorial, serão necessários 3 programas:
- Gromacs: O programa que de fato será utilizado para executar a simulação além de algumas análises
- Travis (apenas para parte 2): Será empregado para a realização de algumas análises
- VMD: Será utilizado para a visualização das estruturas e trajetórias geradas
- Grace: Será utilizado para gerar gráficos dos resultados obtidos
A seguir, discutiremos brevemente a instalação de cada um desses programas.
Gromacs: Pode ser baixado diretamente de muitos repositórios Linux. Verifique o gerenciador de aplicativos do seu sistema. A versão disponível pode ser alguma versão um pouco antiga, mas deverá servir para nossos tutoriais. Pode também fazer o download da versão mais atual ou de outra de sua escolha pelo site https://manual.gromacs.org/current/download.html e instalar seguindo as instruções deles. Para uma instalação simples, basta seguir os passos (necessita o cmake instalado, se não o tiver, verifique o repositório de programas do seu sistema):
1) Descompacte os arquivos de instalação com o comando (no lugar do *, indique a versão):
tar xfz gromacs-*.tar.gz
2) Vá para o diretório descompactado e crie um subdiretório com nome “build”:
cd gromacs-2023
mkdir build
cd build
3) Rode o cmake para configurar a instalação e o comando make para realizá-la (essas etapas podem demorar um pouco):
cmake .. -DGMX_BUILD_OWN_FFTW=ON -DREGRESSIONTEST_DOWNLOAD=ON
make
4) Complete a instalação com os comandos (será necessário ter privilégios de administrador nessa etapa):
sudo make install
source /usr/local/gromacs/bin/GMXRC
Teste sua instalação com o comando:
gmx mdrun -h
Observação: Dependendo da sua instalação, um sufixo pode ser adicionado no comando gmx, tal como gmx_mpi ou gmx_d. Nesse caso, sempre que no tutorial usarmos comandos do gromacs, substitua “gmx” pela versão com o sufixo apropriado.
.
Travis: Esse programa será utilizado apenas na segunda etapa do tutorial. Faça o download do “source code” em http://www.travis-analyzer.de/ . Descompacte ele com o comando tar xfz de modo similar ao indicado acima para o Gromacs. Entre no diretório descompactado e digite o comando make. Se tudo der certo, será criado o diretório exe/ e dentro dele estará o binário do Travis. Pode opcionalmente copiá-lo para o diretório /usr/bin (precisará de permissões de administrador) para poder executá-lo de qualquer diretório com o comando travis.
.
VMD: Faça o download no site https://www.ks.uiuc.edu/Development/Download/download.cgi?PackageName=VMD (será necessário fazer um rápido cadastro). Descompacte o arquivo com tar xfz e entre no diretório com nome vmd seguido da versão. Em seguida, execute o comando
./configure
e, como root,
make install
.
Grace: Esse programa está presente nos repositórios das distribuições linux. Faça a instalação diretamente pelo repositório.
.
2. Criação e visualização da caixa de simulação
Para a realização de uma simulação, devemos primeiro definir uma estrutura inicial, com as coordenadas x, y, z de cada átomo presente no sistema modelo. Para simulações com água, podemos nos valer de uma estrutura de água líquida já pronta e incluída na instalação do próprio Gromacs. Essa é uma caixa cúbica com 216 moléculas de água existente no arquivo spc216.gro. Esse arquivo deve estar localizado no diretório /usr/share/gromacs/top/ ou /usr/local/gromacs/share/gromacs/top/, dependendo da sua instalação do Gromacs. Caso não encontre esses diretórios, pode localizar o arquivo com o comando
locate spc216.gro
Abra o mesmo em um editor de textos como vi, vim ou nano ou mesmo com o comando “more spc216.gro”. Abaixo são mostradas as primeiras linhas desse arquivo:
16H2O,WATJP01,SPC216,SPC-MODEL,300K,BOX(M)=1.86206NM,WFVG,MAR. 1984
648
1SOL OW 1 .230 .628 .113
1SOL HW1 2 .137 .626 .150
1SOL HW2 3 .231 .589 .021
2SOL OW 4 .225 .275 -.866
2SOL HW1 5 .260 .258 -.774
2SOL HW2 6 .137 .230 -.878
A primeira linha de um arquivo .gro é um comentário, que pode ser usada para descrever o sistema ou poderia ser deixada em branco, não sendo importante para a execução da simulação. A segunda linha dá a quantidade total de átomos no sistema e as linhas seguintes descrevem a estrutura de fato. Nessas, a primeira coluna é o número e nome da molécula, a segunda coluna é o nome dado a cada átomo, no caso “OW” para os átomos de oxigênio e “HW1” e “HW2” para os de hidrogênio. A terceira coluna é o número do átomo e entre a quarta e a sexta coluna são dadas as coordenadas x, y e z de cada átomo em nm. Opcionalmente, o arquivo .gro pode incluir ainda mais 3 outras colunas com as componentes x, y e z da velocidade de cada átomo, mas não é o caso desse exemplo. Na última linha do arquivo são dadas as dimensões da caixa de simulação, que é um cubo com arestas 1,86206 nm.
Para esse tutorial, vamos usar uma caixa de simulação maior. Para isso vamos usar o comando gmx solvate, que também será usado nos próximos tutoriais para solvatar outras moléculas. Com esse comando, podemos especificar a caixa de solvente que vamos usar como base com a opção -cs, as dimensões da caixa de simulação em nanômetros com a opção -box (demanda 3 valores, um para cada eixo cartesiano), -o permite especificar o nome do arquivo da saída e -maxsol limita a quantidade máxima de moléculas de solvente a serem inseridas, no caso usaremos 1500. Sem a opção -maxsol, seriam inseridas quantas moléculas de solvente forem possíveis dentro da caixa especificada. A linha de comando completa é mostrada abaixo:
gmx solvate -cs spc216.gro -box 3.7 3.7 3.7 -o conf.gro -maxsol 1500
Verifique o arquivo gerado em um editor de textos. O número total de átomos agora deverá ser 4500.
Abra o arquivo conf.gro com o vmd:
vmd conf.gro
A estrutura deverá carregar com representação similar à mostrada abaixo, com os átomos de oxigênio em vermelho e os de hidrogênio em branco:

No restante dessa sessão, trabalharemos com algumas representações possíveis no VMD, caso já esteja familiarizado com o mesmo, pode avançar para a próxima etapa do tutorial para realização da minimização de energia.
Primeiro, vamos incluir as arestas da caixa de simulação. Para isso, na janela “VMD main”, abra o menu “Extensions” e escolha “Tk console”. Na janela aberta, digite o comando
pbc box -color green
e tecle Enter. Serão incluídas na representação as arestas da caixa de simulação. Ao invés de “green” pode especificar outra cor de sua preferência.
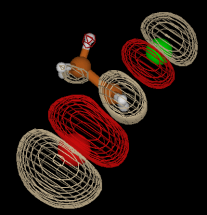
Feito isso, pode fechar a janela “VMD TkConsole”. De volta a janela “VMD main”, pode no menu “Display” alternar entre a representação perspectiva e ortográfica. Em “Graphics” > “Colors” pode alterar cores usadas para representar os átomos escolhendo a categoria “Name” ou do plano de fundo na opção “Background”. Vamos alterar o fundo para branco e os átomos de hidrogênio para cinza. Após isso, clique em “Graphics” > “Representations”. Será aberta uma nova janela. Na opção “Drawing methods”, que está selecionada como “lines” pode alterar para “VDW”, para que os átomos sejam representados com esferas com os respectivos raios de van der Waals. Com essas alterações, a representação gerada deverá se parecer com a mostrada abaixo:

Note que há um número grande de moléculas localizadas nas faces da caixa de simulação. Isso geraria uma tensão superficial elevada pois as moléculas nas faces da caixa sentiriam forças atrativas apenas para o lado de dentro da mesma. Tal situação não é realista para simular uma quantidade macroscópica de líquido onde, mesmo havendo interfaces, o número de moléculas nas mesmas é extremamente pequeno se comparado ao número total de moléculas no líquido. Esse artefato é contornado nas simulações por meio do uso de condições periódicas de contorno, onde a caixa de simulação é rodeada por infinitas réplicas dela mesma. Podemos visualizar as réplicas no VMD clicando na aba “periodic” na janela de “Graphic Representations” e escolhendo em quais dimensões quer mostrar as réplicas, como mostrado para as réplicas nas direções x e y na figura abaixo:

Teste outras representações para a caixa de água! Algumas representações podem não funcionar por serem específicas para biomoléculas (como “ribbons”) ou por demandarem outros dados (como “isosurface”). Duas interessantes são a CPK, que corresponde ao modelo de bola e vareta, e a “Hbonds”, que mostra as ligações de hidrogênio entre as moléculas. Você pode gerar mais de uma representação simultaneamente, é interessante mostrarmos, por exemplo, as ligações de hidrogênio ao mesmo tempo que mostramos também as moléculas. Para isso, deixe uma representação como “CPK”, crie uma nova representação no botão “Create Rep.” e nesse mude o tipo para “Hbonds”. Pode ainda alterar a espessura das linhas usadas para as ligações de hidrogênio nessa janela em “line thickness” e a cor que as mesmas são mostradas alterando o “Coloring method” para “Color ID” e escolhendo qual cor deseja usar. No caso, alterei para laranja para gerar a representação abaixo:

Para fechar o VMD, feche a janela “VMD main”.
.
.
3. Minimização de energia
A estrutura inicial gerada pode conter interações fortemente repulsivas, que podem ocasionar a queda da simulação logo nos primeiros passos ou então artefatos em estruturas mais complexas. Para evitar tais problemas, é interessante antes de realizar a simulação de dinâmica molecular propriamente dita fazermos a minimização de energia. Na minimização, ao invés de serem integradas equações de movimento, são realizados pequenos deslocamentos nas partículas a fim de reduzir a energia potencial do sistema. Com isso, interações fortemente repulsivas são com frequência eliminadas com poucos passos de minimização.
Para executar a minimização, começamos criando um novo diretório e copiaremos a estrutura gerada para ele com os comandos:
mkdir minimize
cp ../conf.gro .
Faça o download dos arquivos grompp_minimize.mdp e topol.top para o diretório criado. O arquivo .top indica os parâmetros de interação ligantes e não-ligantes necessários para descrever cada molécula do sistema. Discutiremos em mais detalhes esse arquivo em um tutorial futuro focado no estudo de moléculas em água.
O arquivo .mdp indica as condições de simulação, incluindo informações como o tipo de cálculo (minimização, dinâmica molecular, etc), número de passos a ser executado, raio de corte e correções de longo alcance para interações não ligantes, uso de condições periódicas de contorno entre outras. Visualize o arquivo grompp_minimize.mdp com algum editor de textos. Inseri comentários iniciados com ; em cada linha explicando o que cada uma representa. Mais detalhes sobre os parâmetros de controle das simulações podem ser vistos nessa página: (em inglês).
Para executar a minimização (e também simulações de dinâmica molecular), devemos fazer primeiro o pré-processamento dos arquivos de input com o comando:
gmx grompp -f grompp_minimize.mdp -maxwarn 2
Serão lidos os arquivos conf.gro, topol.top e grompp_minimize.mdp. Se estiver tudo certo, será gerado o arquivo topol.tpr, que é lido pelo programa mdrun, que de fato realiza a simulação ou, no caso, a minimização de energia. Para realizá-la, use o comando:
gmx mdrun -v
Pode opcionalmente usar -nt (número inteiro) para limitar o número de treads de processamento a serem usadas. Sem essa opção, o programa usará quantas estiverem disponíveis. A opção -v indica que, em uma minimização, ele deve imprimir na tela o passo de simulação, o deslocamento máximo no passo, a energia potencial, a força máxima encontrada e o átomo sujeito à maior força resultante. Compare abaixo os resultados para o primeiro e para o último passo:
Step= 0, Dmax= 5.0e-02 nm, Epot= -5.60041e+04 Fmax= 3.06329e+03, atom= 775
Step= 3000, Dmax= 1.0e-03 nm, Epot= -7.93261e+04 Fmax= 3.79296e+02, atom= 394
Apesar de ter ocorrido uma redução razoável na energia potencial, não houve uma mudança de ordem de grandeza e a maior força logo na estrutura inicial não era particularmente alta. Para uma simulação simples como essa e partindo da replicação de uma caixa já relaxada como fizemos a minimização seria até dispensável. Para sistemas mais complexos ou mesmo estruturas iniciais preparadas sem ser com base em outras já relaxadas, entretanto, a minimização acaba sendo quase sempre necessária antes de realizar a simulação. Então, para fins didáticos e de boas práticas a realizamos aqui.
Serão gerados alguns arquivos de output como o confout.gro, que contém a estrutura final da minimização, e o ener.edr, que inclui a variação de diversas componentes de energia ao longo da minimização. Podemos gerar, por exemplo, o gráfico da variação da energia potencial com o comando:
gmx energy -f ener.edr -o potential
Observação: Na maior parte dos comandos do gromacs, a opção “-f” indica um arquivo a ser lido como input enquanto “-o” define o nome a ser dado a um arquivo de saída. A opção -h faz com que seja mostrada a ajuda do programa ao invés de executar o comando
Ao dar enter, aparecerão as várias componentes de energia calculadas ao longo da simulação. Digite o número da opção correspondente à energia potencial e dê Enter 2 vezes. Será gerado o arquivo potential.xvg. Abra ele com o xmgrace ou outro visualizador de gráficos:
xmgrace potential.xvg
A imagem abaixo mostra a variação de energia potencial ao longo da minimização. Apesar de no eixo horizontal aparecer escrito tempo, no caso de uma minimização o correto seria falar em número de passos pois, diferente da dinâmica molecular, a minimização não descreve a evolução temporal do sistema.

.
.
4. Simulação de dinâmica molecular
Após ter sido realizada a minimização, vamos criar um novo diretório para a execução da simulação de dinâmica molecular. Faremos uma simulação no ensemble NPT, ou seja, com número de partículas, pressão e temperatura constantes, portanto, vamos criar uma pasta com o nome NPT para rodar a simulação. Partindo do diretório onde foi executada a minimização, vamos voltar para o diretório acima e criar uma pasta com o nome NPT usando os comandos no terminal:
cd ..
mkdir NPT
cd NPT
Copie o mesmo arquivo de topologia que usamos na minimização para esse diretório além de copiar a estrutura final da minimização e renomeá-la como “conf.gro”. Essa estrutura será usada como ponto de partida para a simulação:
cp ../minimize/topol.top .
cp ../minimize/confout.gro conf.gro
Além da topologia e do arquivo de estrutura, precisamos também do arquivo grompp.mdp com as opções de controle da simulação. Faça o download dele aqui e o salve no diretório onde irá rodar a simulação. Assim como no caso da minimização, coloquei comentários no arquivo grompp.mdp explicando cada linha. Algumas opções serão discutidas em maiores detalhes a seguir:
Nesse tutorial, realizaremos a simulação na temperatura de 300 K mantida pelo algoritmo V-rescale e pressão de 1 bar mantida pelo acoplamento de Berendsen. A temperatura de um sistema é determinada pela distribuição de velocidades das partículas (vide página sobre a distribuição de Maxwell-Boltzmann), dessa forma, se a temperatura está abaixo da desejada o termostato irá gerar um aumento nas velocidades moleculares. O acoplamento de pressão faz com que o volume da caixa de simulação possa variar, sendo que ela irá se expandir caso a pressão interna seja maior que a externa (1 bar) ou contrair caso contrário.
Outras variáveis de controle importante dizem respeito ao passo de integração dt e ao número total de passos. O passo de integração corresponde ao intervalo de tempo no qual as forças atuantes sobre cada partícula são consideradas constantes a fim de permitir a integração das equações de movimento. Passado esse tempo dt, as forças são recalculadas. Um valor alto de dt leva a instabilidades na simulação pois as forças deveriam ter variado significativamente dentro de um intervalo de tempo onde foram consideradas constantes. Tais instabilidades podem resultar em flutuações grandes de temperatura ou mesmo levar a simulação a cair por surgirem forças repulsivas muito intensas. Por outro lado, um valor muito baixo de dt implica que um número muito grande de passos será necessário para atingir o mesmo tempo, ou seja, será necessário mais tempo de cálculo para observar um mesmo fenômeno. Uma boa regra diz que o valor ótimo de dt é 10% do período do movimento mais rápido esperado no sistema. Em sistemas que possuem átomos de hidrogênio, como é o caso da simulação de água, o movimento mais rápido (maior frequência) é o estiramento das ligações covalentes envolvendo os átomos de hidrogênio. Nesse caso, o passo de integração recomendado é de 1 fs (1 femtossegundo). Em sistemas envolvendo apenas átomos mais pesados (como uma simulação de um cristal de NaCl) ou com modelos de átomos unidos ou modelos coarse grained, onde átomos mais leves são agrupados em sítios de interação de maior massa, valores maiores de passo de integração podem ser usados.
Para esse tutorial, faremos uma simulação de 1 ns com passo de integração de 1 fs. O total de passos é dado pela razão entre o tempo desejado (1000 ps) e o passo de integração (0,001 ps), no caso é então 1 milhão de passos.
Assim como no caso da minimização, devemos primeiro fazer o pré-processamento dos arquivos de input com o comando:
gmx grompp -maxwarn 2
Se estiver tudo certo, será gerado o arquivo topol.tpr. Será impressa na tela uma estimativa do tamanho dos outputs gerados (65 Mb). Execute agora a simulação com o comando:
gmx mdrun -v
Será atualizada na tela constantemente o horário previsto para o término da simulação. Essa etapa pode levar algumas horas. Caso esteja levando muito tempo, reduza o número de passos no arquivo grompp.mdp e execute novamente o comando gmx grompp. Menos passos indica pior relação sinal ruído nas análises e pode não ser suficiente para a relaxação do sistema, mas para efeitos de realização do tutorial pode trabalhar com um número de passos menor.
A trajetória gerada pode ser visualizada com o VMD digitando:
vmd conf.gro traj_comp.xtc
Observação: Não é possível abrir diretamente a trajetória em formato xtc no vmd, sendo necessário carregar primeiro um arquivo de estrutura em outro formato como o conf.gro.
Note que a visualização da trajetória ficará bem estranha, com “ligações” do tamanho da caixa de simulação. Isso é um artefato criado na representação gráfica quando moléculas são “quebradas”, com alguns átomos estando próximos a uma extremidade da caixa enquanto outros estão próximos à extremidade oposta. Tais moléculas aparentemente quebradas são apenas um artefato de visualização, pois, devido às condições periódicas de contorno, os átomos que aparecem em extremidades opostas ainda estão em uma distância compatível com as ligações químicas. Pode gerar uma nova trajetória com as moléculas aparecendo sempre inteiras com o comando:
gmx trjconv -f traj_comp.xtc -pbc mol -o traj_whole.xtc
Escolha gerar uma nova trajetória com todo o sistema digitando zero e dando Enter. Visualize o novo arquivo de trajetória no VMD:
vmd conf.gro traj_comp.xtc
Na próxima página trabalharemos com algumas análises usando ferramentas do próprio Gromacs e também do programa Travis.
.
.
5. Receita de bolo (tutorial sem explicações):
Prepare a caixa de simulação com 1500 moléculas de água:
gmx solvate -cs spc216.gro -box 3.7 3.7 3.7 -o conf.gro -maxsol 1500
Crie um diretório para a minimização de energia, copie o conf.gro para ele bem como os arquivos topol.top e grompp_minimize.mdp disponíveis para download. Faça o pré-processamento dos arquivos de input e execute a minimização:
gmx grompp -f grompp_minimize.mdp -maxwarn 2
gmx mdrun -v
Calcule a variação de energia potencial na minimização e a visualize com o Grace (no primeiro comando, digite o número correspondente a opção “Potential” e dê Enter 2 vezes):
gmx energy -f ener.edr -o potential
xmgrace potential.xvg
Crie um novo diretório para a realização da simulação de dinâmica molecular. Copie os arquivos topol.top e confout.gro da minimização e o renomeie o segundo para conf.gro. Faça o download do arquivo grompp.mdp para a mesma pasta e execute os comandos:
gmx grompp -maxwarn 2
gmx mdrun -v
Quando a simulação terminar, visualize a trajetória no VMD:
vmd conf.gro traj_comp.xtc
Gere um novo arquivo trajetória tornando as moléculas nas beiradas da caixa inteiras:
gmx trjconv -f traj_comp.xtc -pbc mol -o traj_whole.xtc
Visualize o novo arquivo de trajetória no VMD:
vmd conf.gro traj_comp.xtc
.